针对肿瘤组织的SmallRNA(一类大小约17~40个碱基的单链小分子RNA,主要包括miRNA,snoRNA,piRNA等),通过新一代高通量测序获得样本细胞中小RNA的序列信息,表达水平及其表达差异。
注:提取基因组DNA,利用Covaris进行随机打断,电泳回收所需长度的DNA片段(0.2~5Kb),加上接头,进行cluster制备(Solexa)或E-PCR(SOLiD),最后利用Paired-End(Solexa)或者Mate-Pair(SOLiD)的方法对插入片段进行重测序。
服务流程:
测序深度(SequencingDepth):测序得到的碱基总量(bp)与基因组大小(Genome)的比值,它是评价测序量的指标之一。测序深度与基因组覆盖度之间是一个正相关的关系,测序带来的错误率或假阳性结果会随着测序深度的提升而下降。重测序的个体,如果采用的是双末端或Mate-Pair方案,当测序深度在10~15X以上时,基因组覆盖度和测序错误率控制均得以保证。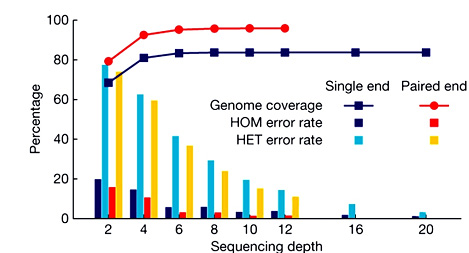
测序深度对基因组覆盖度和测序错误率的影响
(HOM:纯合体HET:杂合体)
全基因组重测序的个体,通过序列比对,可以找到大量的单核苷酸多态性(SNP),插入缺失(InDel,Insertion/Deletion)和结构变异(SV,StructureVariation)位点。SBC可以协助客户,通过生物信息手段,分析不同个体基因组间的结构差异,同时完成SNP及基因组结构注释。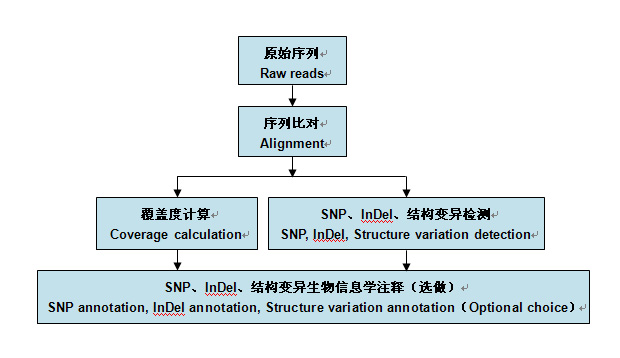
全基因组重测序生物信息学分析流程
1.数据量产出
总碱基数量、TotalMappingReads、UniquelyMappingReads统计,测序深度分析。
2.一致性序列组装
与参考基因组序列(Referencegenomesequence)的比对分析,利用贝叶斯统计模型检测出每个碱基位点的最大可能性基因型,并组装出该个体基因组的一致序列。
3.SNP检测及在基因组中的分布
提取全基因组中所有多态性位点,结合质量值、测序深度、重复性等因素作进一步的过滤筛选,最终得到可信度高的SNP数据集。并根据参考基因组信息对检测到的变异进行注释。
4.InDel检测及在基因组的分布
在进行mapping的过程中,进行容gap的比对并检测可信的shortInDel。在检测过程中,gap的长度为1~5个碱基。对于每个InDel的检测,至少需要3个Paired-End序列的支持。
5.StructureVariation检测及在基因组中的分布
目前SBC能够检测到的结构变异类型主要有:插入、缺失、复制、倒位、易位等。根据测序个体序列与参考基因组序列比对分析结果,检测全基因组水平的结构变异并对检测到的变异进行注释。







